Analizando junto a @lorenz_efe los primeros genomas de SARS-COV2 de Argentina podemos decir algunas cosas:
Analizando junto a @lorenz_efe los primeros genomas de SARS-COV2 de Argentina podemos decir algunas cosas:.
los 3 genomas muestran gran similitud con otros provenientes de Europa, USA y Asia, y junto a que tienen algunas pocas diferencias entre sí, nos hace pensar que
estas 3 cepas del virus corresponden a 3 introducciones diferentes en el país, es decir que los virus aislados de los 3 pacientes vinieron de diferentes lugares. La plataforma @nextstrain (un gran grupo de trabajo) toma los genomas del repositorio global (@GISAID) y realiza el análisis en base a la secuencia genómica, la localización geográfica, la fecha en que se aisló de un paciente, etc. Y muestra casi en tiempo real la comparativa global. (nuestros genomas aparecieron horas después de subirlos!!)
Yendo un poquito más en profundidad, y gracias al gran movimiento open access en torno a la pandemia, podemos decir que según una de las clasificaciones (@GISAID) sobre los SARS-COV2 los 3 pertenecerían al mismo grupo denominado G (que se está transformando en el mayoritario).
Tiene esto alguna implicancia en, por ejemplo, la virulencia (capacidad del virus de producir la enfermedad y la severidad asociada)? ?
Por el momento no hay evidencia o estudio concluyente sobre este tema ?. Es más, van apareciendo diferentes clasificaciones para ver si se puede concluir algo en base a los agrupamientos que se observan al comparar los genomas obtenidos en todo el mundo.
Hace un par de días salió un artículo @arambaut y su grupo (que la rompe en el análisis genómico de virus) que propone una clasificación dinámica, que nos permita crear la clasificación a medida que vamos obteniendo nuevos genomas. Por qué es importante?
http://virological.org/t/a-dynamic-nomenclature-for-sars-cov-2-to-assist-genomic-epidemiology/458 …
Es muy importante para este tipo de virus de ARN que suelen cambiar «bastante» en el tiempo como explica el Dr. Kornblihtt en la nota en pagina 12, www.pagina12.com.ar/258224-secuencias-y-consecuencias-de-los-virus ….
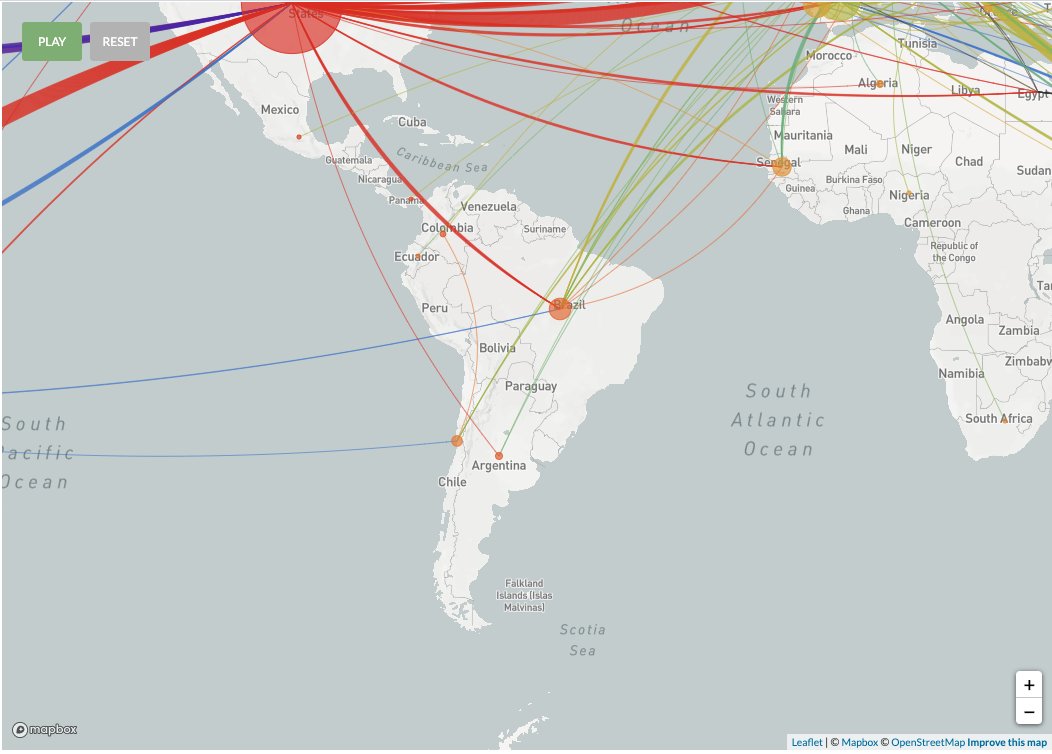
En base a sus análisis, como explica @silargi , podemos concluir, con cierta confianza, que los 3 virus secuenciados de Argentina provienen 1 de USA y los otros 2 de Europa, y los 3 pertenecen al mismo linaje B1 según esta última clasificación.
Hoy mirando de nuevo el mapa de @nextstrain vi que actualizaron las transmisiones inferidas en base a los genomas argentinos de COVID-19 que produjo el @ANLIS_Malbran. Ahora indican dos introducciones desde Europa y una desde Norteamérica.
Es tan increíble el laburo a nivel global que se está haciendo, que día a día cambia el resultado del análisis en base al aumento del número de genomas, por esto es crucial que todas las regiones estén bien representadas en número, por lo cual tenemos que seguir secuenciando!!
Pero vamos a las preguntas de interés público
Para que sirve secuenciar el genoma del coronavirus??
En ésta nota la Dra. Perandones comunica un poco la idea de para qué sirve conocer el genoma de los virus que circulan en nuestro país.
www.pagina12.com.ar/258117-coronavirus-en-argentina-lograron-descifrar-el-genoma-del-co …
En resumen, esto tiene impacto en la salud pública y las estrategias para frenar el avance del virus, en el diagnóstico, en la vacuna (lograr que los virus que circulan en Argentina estén representados en la formulación vacunal).
La secuenciación comprendió muchos actores en el camino desde la toma de muestra hasta el análisis, y quiero remarcar a los directivos de @ANLIS_Malbran, al Departamento de Virología y especialmente a todos en el Servicio de Virosis Respiratorias que se están rompiendo el alma.
Es una lista muy grande para nombrar en el hilo, pero no me quiero olvidar de nombrar a Elsa Baumeister (jefa de Virosis Respiratorias), Josefina Campos (jefa de la Plataforma de Genómica y Bioinformática) y Daniel Cisterna (Depto. de Virología)
este logro fue posible y seguirá dando sus frutos por el esfuerzo conjunto de todo el @ANLIS_Malbran (y todo el sistema de salud), todos los profesionales de la salud del Malbran estamos a disposición, contribuyendo de las formas más variadas a las tareas referidas a la pandemia.
E incluyo además el esfuerzo que está haciendo el sistema de salud de Argentina completo, médicos, enfermeros, bioquímicos, personal de salud, científicos, funcionarios del @msalnacion poniendo el cerebro y el cuerpo al servicio de la salud en Argentina.
Gracias a todos!!